
寻找同源蛋白家族用的比较多的是 Orthomcl,但是该软件多年前已经停止更新,且使用的时候需要安装和使用 MySQL,操作起来比较繁琐。因此 OrthoFinder 应运而生,并且更新到版本 2。后者不但可以寻找同源家族,并且可以构建基因家族进化树。
- OrthoFinder 及依赖包的安装
- 下载
wget https://github.com/davidemms/OrthoFinder/releases/download/v2.2.7/OrthoFinder-2.2.7.tar.gz |
- 解压
tar zxvf OrthoFinder-2.2.7.tar.gz |
- 安装(加入环境变量即可)
vim ~/.bashrc | |
i | |
export PATH=$PATH:$HOME/tools/OrthoFinder-2.2.7 | |
Esc | |
shift + ; | |
wq! | |
source ~/.bashrc |
依赖包
(1)DIAMOND
下载对应版本,解压并将主程序拷贝至存在于环境变量的目录下或将其所在的目录加入环境变量:
wget https://github.com/bbuchfink/diamond/releases/download/v0.9.24/diamond-linux64.tar.gz | |
tar xzf diamond-linux64.tar.gz | |
sudo cp diamond /usr/local/bin |
没有 root 权限的可以把 diamond 所在目录加入环境变量。
(2) MMseqs2
下载对应版本,解压并将主程序拷贝至存在于环境变量的目录下或将其所在的目录加入环境变量:
wget https://github.com/soedinglab/MMseqs2/releases/download/7-4e23d/MMseqs2-Linux-AVX2.tar.gz | |
tar xzf MMseqs2-Linux-AVX2.tar.gz | |
sudo cp mmseqs2/bin/mmseqs /usr/local/bin |
(3) MCL
- Ubuntu, Debian, Linux Mint 安装方法:
sudo apt-get install mcl |
- Centos, Redhat 安装方法:
wget https://micans.org/mcl/src/mcl-latest.tar.gz | |
tar zxvf mcl-latest.tar.gz | |
cd mcl-14-137(视具体情况而定) | |
./configure | |
make | |
make check | |
sudo make install |
(4) FastME
下载二进制文件,解压并将主程序拷贝至存在于环境变量的目录下或将其所在的目录加入环境变量:
wget http://www.atgc-montpellier.fr/download/sources/fastme/fastme-2.1.5.tar.gz | |
tar zxvf fastme-2.1.5.tar.gz | |
sudo cp fastme-2.1.5/binaries/fastme-2.1.5-linux64 /usr/local/bin/fastme |
(5) 可选: BLAST+
- Ubuntu, Debian, Linux Mint 安装方法:
sudo apt-get install ncbi-blast+ |
- Centos, Redhat 安装方法:
wget ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.8.1+-x64-linux.tar.gz | |
tar zxvf ncbi-blast-2.8.1+-x64-linux.tar.gz | |
vim ~/.bashrc | |
i | |
export PATH=$PATH:$HOME/tools/ncbi-blast-2.8.1+/bin | |
Esc | |
shift + ; | |
wq! | |
source ~/.bashrc |
- 运行 OrthoFinder
(1) 运行示例数据:
cd OrthoFinder-2.2.7 | |
orthofinder -f ExampleData |
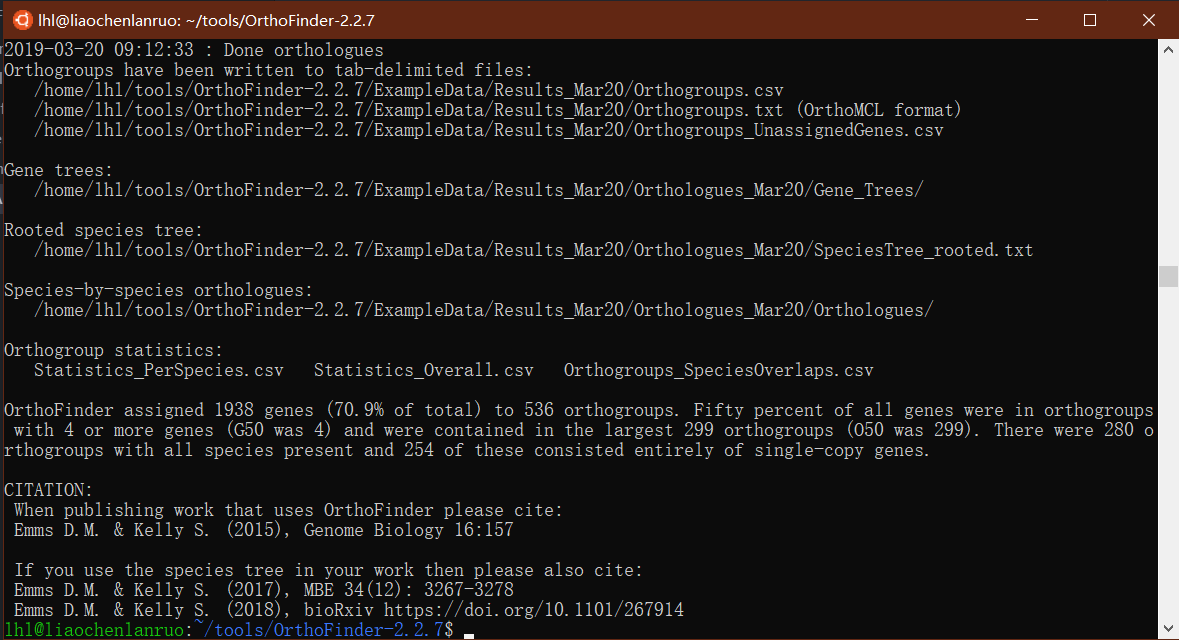
运行结果如下,会显示输出文件的路径,表明运行成功:
(2) 运行自己的数据:
- step1:数据准备
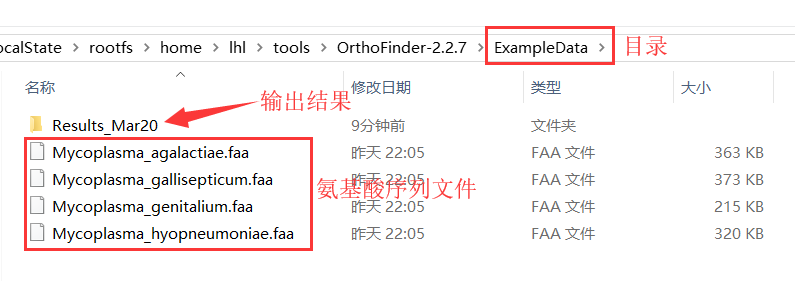
下载氨基酸序列,要求为 fasta 格式,每个物种一个文件。将所有 fasta 文件存于一个目录中(如 Data 目录),如下图所示。文件名要简洁并有区分性,因为文件名会作为最终的物种 ID。
- step2:运行程序
在 Data 目录的上一级目录打开终端,运行如下命令:
orthofinder -f Data -t 线程数 |
- 结果解读
(1) Results Files: Orthogroups
包含一个主文件 “Orthogroups.csv” 和两个支持文件:
Orthogroups.csv,每一行为一个 group,每一列为一个物种,行列交汇处为基因名称。
Orthogroups_UnassignedGenes.csv,包含所有未分配到任何 group 的基因名称。
Orthogroups.txt,OrthoMCL 格式的输出结果,内容等同于 Orthogroups.csv。
(2)Results Files: Orthogroup Statistics
包含一些统计数据,可用于比较基因组分析、绘图以及质控。
Statistics_Overall.csv 和 Statistics_PerSpecies.csv,提供基本的描述信息
Orthogroups_SpeciesOverlaps.csv,两两物种的 group 共享矩阵
Species-specific orthogroup:该 group 仅包含一个物种的基因。
G50:group 中的基因数,使得 50%的基因处于该大小或更大的 group 中。
O50:最小数量的 group,使得 50%的基因处于该大小或更大的 group 中。
Single-copy orthogroup:每个物种中只有一个基因的 group(相当于单拷贝核心基因)。这些 group 是构建物种树和许多其他分析的理想选择。
Unassigned gene:未与任何其他基因划分到一个 group 的基因。
(3) Results Files: Orthologues
两两物种间的直系同源基因,每一行为一个 group,第一列为 group 编号,第二列为第一个物种的基因,第三列为第二个物种的基因。同一物种的基因名以 “,” 分割。直向同源物可以是一对一,一对多或多对多。
(4) Results Files: Gene Trees and Species Tree
每个 group 的基因树和定根的物种树以 newick 格式输出,可以用各种看树软件展示,如 MEGA、iTOL、Dendroscope 和 FigTree 等,个人推荐用 iTOL。
- 高级用法
(1)添加新物种到之前的分析
(previous_orthofinder_directory 指的是包含 “SpeciesIDs.txt” 的目录)
orthofinder -b previous_orthofinder_directory -f new_fasta_directory |
(2)从之前的分析中移除物种
从输出目录下找到工作目录 “WorkingDirectory” 中的 “SpeciesIDs.txt” 文件,在要移除的物种那一行最前面加上一个 “#” 并保存,然后运行(previous_orthofinder_directory 指的是包含 “SpeciesIDs.txt” 的目录):
orthofinder -b previous_orthofinder_directory |
(3)同时添加和删除物种
编辑好 “SpeciesIDs.txt” 后,运行:
orthofinder -b previous_orthofinder_directory -f new_fasta_directory |
(4)更多高级功能请阅读官方文档
主要包括 “Inferring MSA Gene Trees”、并行计算、单独运行 BLAST、使用预先计算的 BLAST 结果以及回归检测。
